Estudo liderado por pesquisadores do IFSC/USP alerta para nova ameaça à saúde pública
Créditos – “First Post”
Um estudo inovador liderado por pesquisadores do IFSC/USP, em parceria com o University Medical Center Utrecht (Holanda) e o Hospital Risoleta Tolentino Neves (MG), revelou que um tipo de material genético, chamado plasmídeo IncQ1, está ajudando a espalhar genes que resistem a antibióticos em um hospital brasileiro.
Publicado na prestigiada revista científica Microorganisms, o estudo alerta para o risco crescente desse fenômeno invadir e permanecer no ambiente hospitalar, o que pode dificultar o tratamento de infecções.
Os antibióticos carbapenêmicos são considerados uma das últimas opções para tratar infecções graves, mas, no entanto, muitas bactérias desenvolveram um mecanismo de defesa contra eles, chamado KPC, que torna esses antibióticos ineficazes. O estudo analisou 49 amostras de bactérias coletadas entre 2009 e 2016 em um hospital de Belo Horizonte e descobriu que 67% delas possuíam o gene blaKPC, responsável por essa resistência. Dessas, 85% tinham o gene armazenado em uma configuração genética denominada NTEKPC-IId diferente das normalmente encontradas no país, um pequeno pedaço de DNA que pode ser transferido facilmente entre diferentes bactérias. O grupo identificou, também, que essa configuração genética estava sendo carreada entre as diferentes bactérias do hospital através de um elemento genético conhecido como plasmídeo IncQ1.
Os pesquisadores descobriram que esse material genético pode passar de uma bactéria para outra, atingindo espécies como Klebsiella pneumoniae, Escherichia coli e Enterobacter spp., sem prejudicar a sobrevivência das bactérias, significando que a resistência pode se espalhar rapidamente entre microrganismos diferentes dentro do hospital.
Uma das descobertas mais preocupantes foi a identificação de uma bactéria da espécie Enterobacter cloacae, que possuía 8 plasmídeos diferentes, sendo que quatro deles carregavam um total de 24 genes de resistência a antibióticos de diferentes classes. Além do gene de resistência ao carbapenêmico nesta bactéria, os pesquisadores ressaltam outro gene chamado mcr-9, que confere resistência à colistina – um antibiótico usado como última alternativa para tratar infecções graves. A combinação desses vinte e quatro genes de resistência dificulta ainda mais o tratamento, tornando algumas infecções praticamente intratáveis.
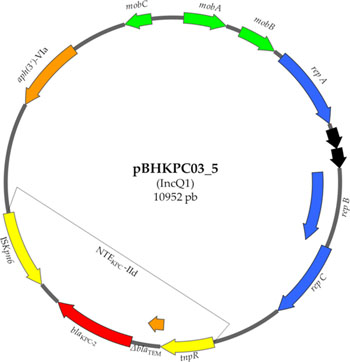
Esquema do plasmídeo IncQ1 que carrega o elemento genético NTEKPC-IId, que se espalhou pelas bactérias do hospital
Os pesquisadores destacam a importância de monitorar e entender como essas bactérias resistentes se espalham para desenvolver estratégias eficazes de combate. “A presença desses plasmídeos móveis e sua capacidade de transmissão entre diferentes espécies representam um grande desafio para o controle da resistência aos antibióticos”, afirma a Profª Dra. Ilana Camargo, do Laboratório de Epidemiologia e Microbiologia Moleculares – LEMiMo – do IFSC/USP e coordenadora do estudo, que acrescenta: “Quando falamos sobre a disseminação da resistência antimicrobiana (RAM), pensamos na disseminação de uma espécie de bactéria resistente por toda a enfermaria do hospital ou UTI. No entanto, esta não é a única maneira pela qual a RAM se espalha”, explica a pesquisadora.

Profa Dra. Ilana Camargo
Neste estudo, conduzido pela Dra. Camila Boralli durante o seu Doutorado junto ao LEMiMo (ISFC-USP), ela mostrou que elementos genéticos móveis podem se esconder em várias espécies de bactérias e dificultar a percepção de que há uma disseminação. “A Dra. Boralli queria estudar diferentes ambientes genéticos carregando o gene blaKPC em bactérias gram-negativas de um hospital em Belo Horizonte. O hospital forneceu bactérias resistentes a carbapenêmicos de 2009 a 2016, e Camila as rastreou para a presença do gene KPC, a presença de Tn4401 e clonalidade. Ela investigou apenas diferentes clones carregando NTEKPC e, para nossa surpresa, havia apenas o tipo NTEKPC-IId naquele hospital. Além disso, esse elemento estava em plasmídeos do tipo IncQ1, mostrando disseminação multiespécies. Embora ela também tenha investigado a capacidade de replicação e conjugação desses plasmídeos em diferentes espécies – cientistas, vale a pena conferir! -, a primeira mensagem que precisamos que você se lembre é que a disseminação de RAM também significa disseminação de genes. Você pode não ter apenas uma espécie se espalhando, mas um plasmídeo escondido e conjugando-se a diferentes espécies bacterianas. Neste caso, plasmídeos do tipo IncQ1 carregando NTEKPC-IId foram encontrados em várias bactérias, incluindo as linhagens clonais de alto risco K. pneumoniae ST11 e ST147”, finaliza a pesquisadora.
Este estudo contou com os financiamentos da CAPES e CNPq.
Para conferir o artigo científico, clique AQUI.
(Créditos da imagem na Home – “Global Press Journal”)
Rui Sintra – Assessoria de Comunicação – IFSC/USP



